
“数学家在咖啡杯和甜甜圈之间看到了同伦,UMAP 在高维数据和二维流形之间看到了同构。”
t-SNE 统治了数据可视化界很多年,直到 2018 年 McInnes 等人提出了 UMAP。
UMAP (Uniform Manifold Approximation and Projection) 建立在深奥的代数拓扑和黎曼几何理论之上,但它的效果却是立竿见影的:
它比 t-SNE 快得多,且保留了更多的全局结构。
在实际的文本分析项目中(如 BERT/OpenAI Embedding),UMAP 已经逐渐成为首选的降维引擎,正是看中了它在速度和结构保持上的平衡能力。
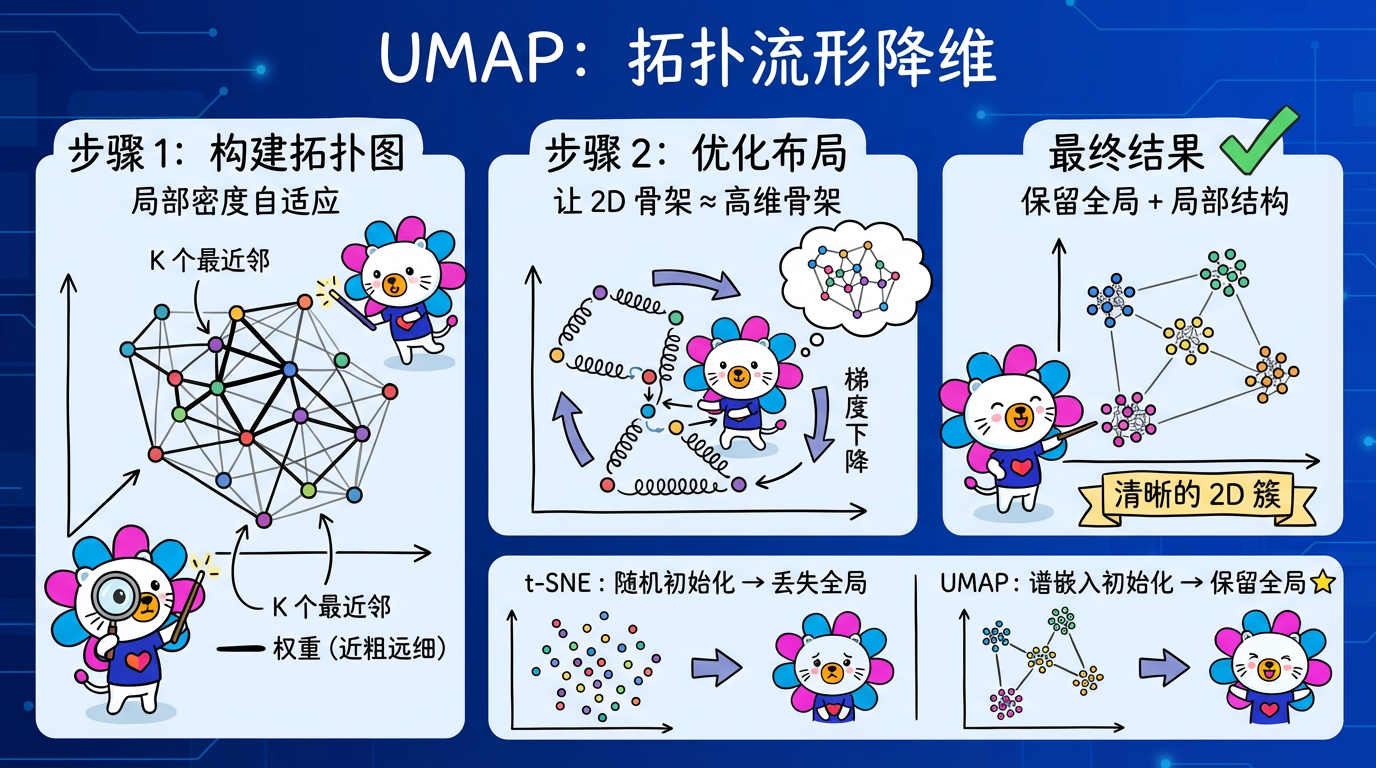
(图注:UMAP 像搭建脚手架一样构建数据的拓扑骨架,然后把它压扁到二维平面。)
1. 核心概念:模糊单纯复形 (Fuzzy Simplicial Complex)
别被这个吓人的名字劝退。我们用人话讲一遍它的核心假设。
1.1 黎曼几何假设:距离是相对的
UMAP 假设数据均匀分布在一个黎曼流形上。
什么是黎曼流形?就是一个局部看起来像欧氏空间(平的),但整体可能弯曲的空间。
关键点在于:在这个流形上,距离也是“局部”的。
- 对于拥挤的点 A:周围全是人。那么距离它 1 米的点 B 就在“旁边”。
- 对于孤独的点 C:位于稀疏区域。那么距离它 10 米的点 D 也算“旁边”,因为周围实在没人了。
UMAP 会根据每个点的局部密度,自动伸缩“尺子”的长度。这使得它能同时处理稀疏和稠密的数据区域。
1.2 算法步骤:脚手架工程
- 构建高维骨架 (Topological Representation):
- 对于每个点,找到它的 K 个最近邻。
- 根据局部密度,赋予每条边一个权重(概率)。密度大的地方,距离单位小;密度小的地方,距离单位大。
- 这就建立了一个加权图(Fuzzy Simplicial Complex)。这就像给数据搭了一个骨架。
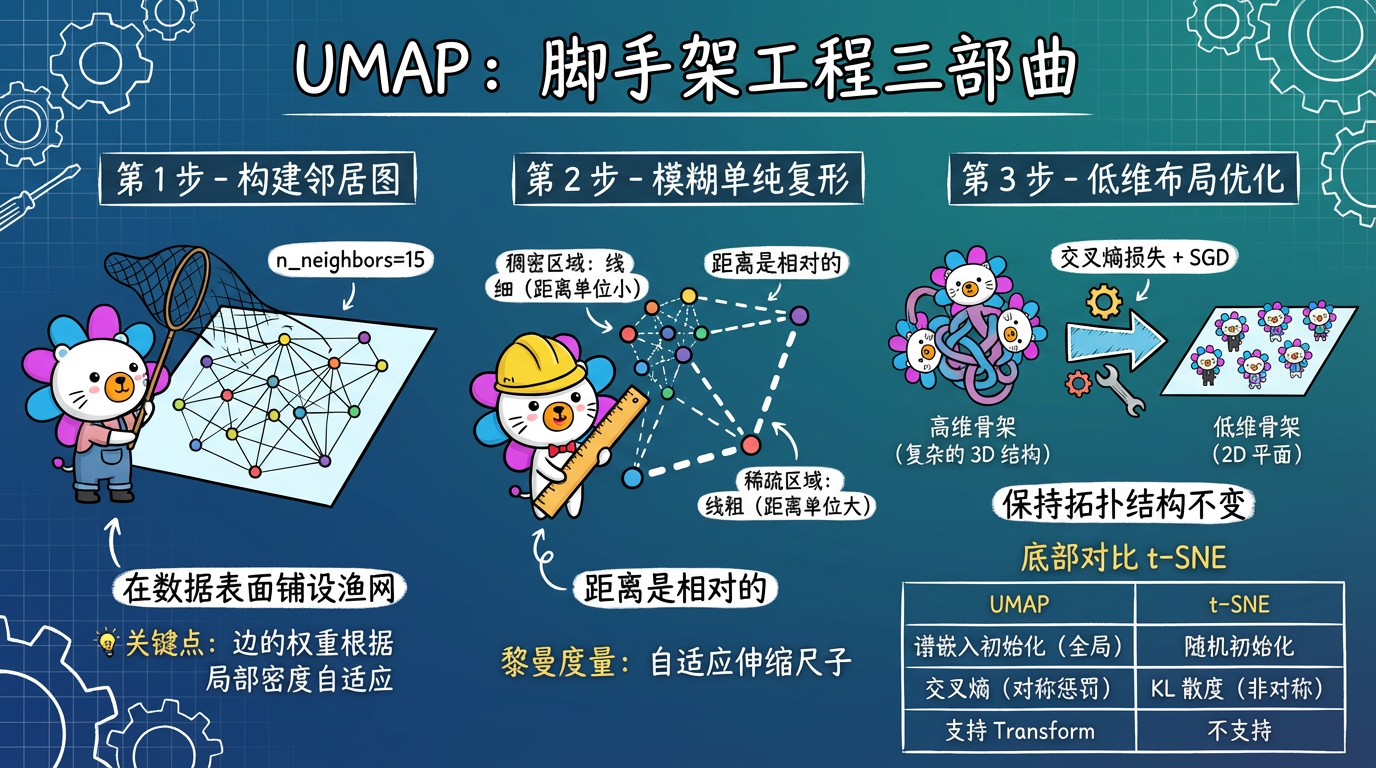
(图注:构建邻居图 → 模糊单纯复形 → 低维布局优化。三步完成拓扑保持降维。)
- 低维布局 (Optimization):
- 在 2D 空间初始化一些点(通常用谱嵌入 Spectral Embedding,这很重要,保留了全局结构)。
- 使用 Cross-Entropy(交叉熵) 作为损失函数,强迫 2D 点的拓扑结构尽可能接近高维骨架。
- 使用随机梯度下降 (SGD) 优化位置。
2. 技术对比:t-SNE vs UMAP
| 特性 | t-SNE | UMAP | 胜出者 |
|---|---|---|---|
| 理论基础 | 概率分布 (KL散度) | 代数拓扑 + 黎曼几何 | UMAP (更坚实) |
| 全局结构 | 丢失 (簇间距离无意义) | 保留较好 (簇间距离有意义) | UMAP |
| 计算速度 | 慢 (尽管有加速) | 快 (快 10-100 倍) | UMAP |
| 初始化 | 随机 / PCA | 谱嵌入 (Spectral) | UMAP (确定性更好) |
| 可扩展性 | 只能可视化 (2D/3D) | 任意维度 (可用于聚类前置) | UMAP |
| 新数据 | 不支持 Transform | 支持 Transform | UMAP (杀手锏) |
为什么 UMAP 能保留全局结构?
- 初始化:UMAP 使用谱嵌入初始化,这本身就是一种保留全局结构的降维方法。
- 损失函数:t-SNE 的 KL 散度只惩罚“该近的不近”,不太惩罚“该远的不远”。而 UMAP 的交叉熵损失对两者都有惩罚,迫使原本离得远的簇在低维空间也得保持距离。

(图注:UMAP 保留全局结构、速度更快、支持新数据 Transform,综合表现优于 t-SNE。)
3. 代码实战:UMAP 的完整流程
在文本分析项目中,UMAP 通常是整个分析链路的枢纽:Embedding -> UMAP -> HDBSCAN。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
import umap
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_digits
# 1. 准备数据
digits = load_digits()
embeddings = digits.data # 64 维
# 2. UMAP 降维
# n_components=2: 降到 2 维用于画图
# n_neighbors=15: 关注周围 15 个邻居(平衡点)
# min_dist=0.1: 点之间至少隔 0.1(防止重叠)
# metric='euclidean': 图像数据用欧氏距离;如果是文本 Embedding,请务必用 'cosine'
reducer = umap.UMAP(
n_components=2,
n_neighbors=15,
min_dist=0.1,
metric='euclidean',
random_state=42
)
coords_2d = reducer.fit_transform(embeddings)
# 3. 绘图
plt.scatter(coords_2d[:, 0], coords_2d[:, 1], c=digits.target, cmap='tab10', s=5)
plt.title('UMAP projection of Digits')
plt.show()
# 4. 杀手锏:处理新数据
# t-SNE 做不到这点!但在生产环境中,我们需要对新来的用户工单进行降维
new_data = np.random.rand(1, 64)
new_coord = reducer.transform(new_data)
print(f"新数据的坐标: {new_coord}")4. 关键参数调优指南
UMAP 虽然好用,但也不是完全免调参的。它有两个参数如同太极的阴阳:
4.1 n_neighbors (邻居数) —— 平衡 局部 vs 全局
- 含义:构建脚手架时,每个点连几根线。
- 小 (2-10):微观视角。关注极局部的细节。图会碎成很多小块,展示数据的精细纹理。
- 大 (50-100):宏观视角。关注全局概貌。图会连成一片,保留大类之间的拓扑关系。
- 推荐:默认值 15 是一个很好的折中。
4.2 min_dist (最小距离) —— 平衡 紧凑 vs 均匀
- 含义:在低维空间,允许点之间挤得多紧。
- 小 (0.0 - 0.1):聚类视角。点紧紧抱团,簇与簇分界清晰。适合后续接 HDBSCAN 聚类。
- 大 (0.5 - 0.99):展示视角。点散得很开,分布均匀,像一张平铺的地图。适合观察整体分布。
- 推荐:0.1 是默认值,适合大多数情况。
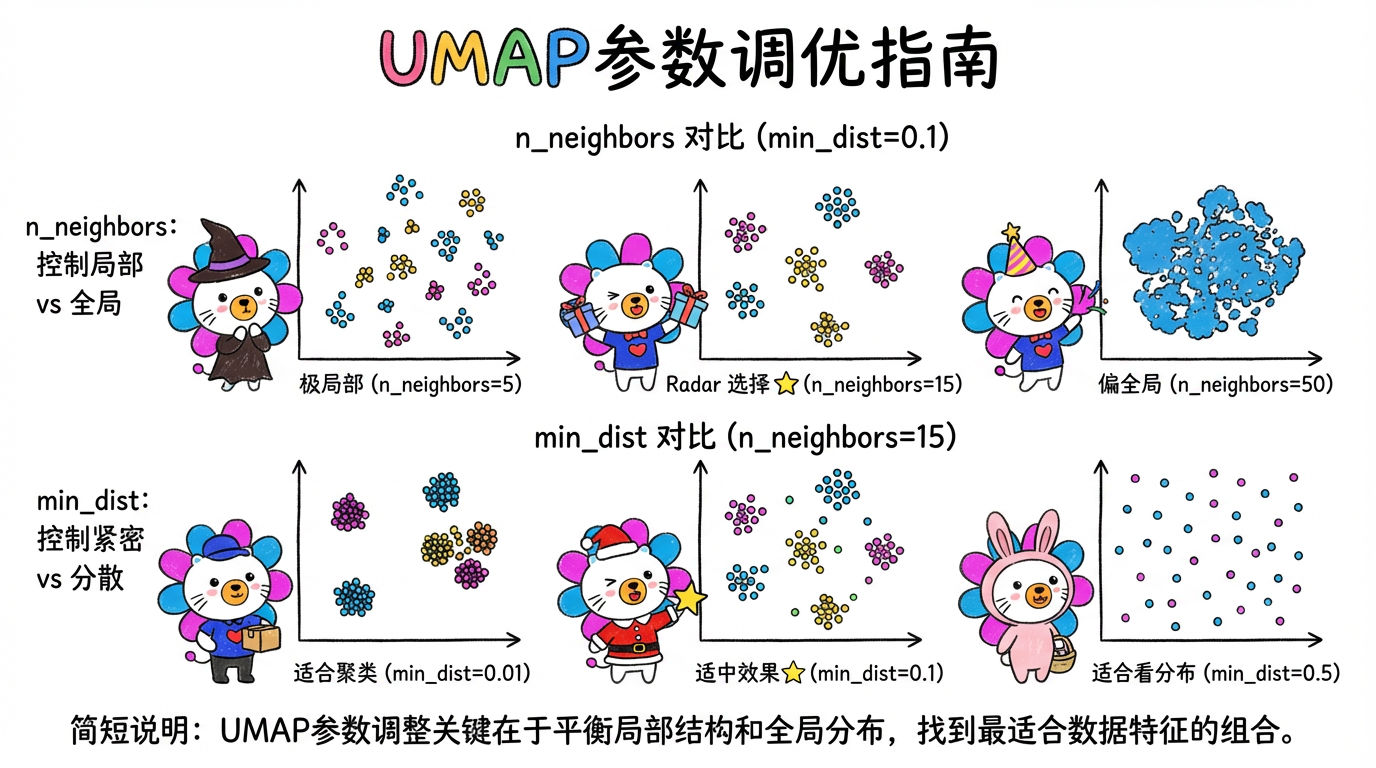
(图注:横轴 min_dist 变大导致点变散;纵轴 n_neighbors 变大导致全局结构更清晰。)
5. 实践要点
- 随机性控制:UMAP 使用随机梯度下降。为了结果可复现,务必固定
random_state=42。 - 不仅是 2D:UMAP 可以降到任意维度。
- 降维策略:如果你要做聚类,强烈建议先用 UMAP 把 1536 维降到 10-50 维,然后再跑 HDBSCAN。这通常比直接在 1536 维上跑聚类效果好得多,也快得多。
- 度量选择:再次强调,对于 BERT/OpenAI 等文本 Embedding 数据,
metric='cosine'是必须的。默认的'euclidean'在高维空间意义不大。
下一章预告
至此,我们的“探索”之旅(聚类+降维)告一段落。
我们手里已经有了锋利的武器:UMAP 帮我们看清结构,HDBSCAN 帮我们发现群体。
从下一章开始,我们将进入更具实战意义的领域——异常检测 (Anomaly Detection)。
如何从数万条看似正常的工单中,揪出那一两条预示着危机的“黑天鹅”?
这不是找“大多数”,而是找“极少数”。





